Windows上にWindows Subsystem for Linux(WSL)を使ってUbuntuを起動し、RStudio Serverを構築して、CmdStanRを使う方法を紹介します。
目次
そもそものきっかけ
現在のStanの計算は、WindowsよりLinuxの方が3倍ぐらい速いみたい(モデルにもよるけど)。Windowsの人はWSLをインストールして実行せよ、そしてRStudioサーバを使って開発するといいよ、とのこと。 https://t.co/6dpU7USlOh
— Kentaro Matsuura (@hankagosa) April 25, 2020
このツイートを教えてもらったのがきっかけ。どうやらWindowsよりもLinuxの方が3倍くらい速いとのこと。
しかし、誰もがLinuxをインストールしたPCを持っているかというとそうでもなく、デュアルブートするのも現実的ではありません。そこで、Windows Subsystem for Linux(WSL)を利用して、Windows上にLinux環境を構築してUbuntuを利用しようというのが主な目的。
簡単にまとめると、仮想マシンでUbuntuを動作させるよりも、WSLの方が動作が軽く良いらしいです。
WSLについての詳細は以下を参照
WSLのインストール
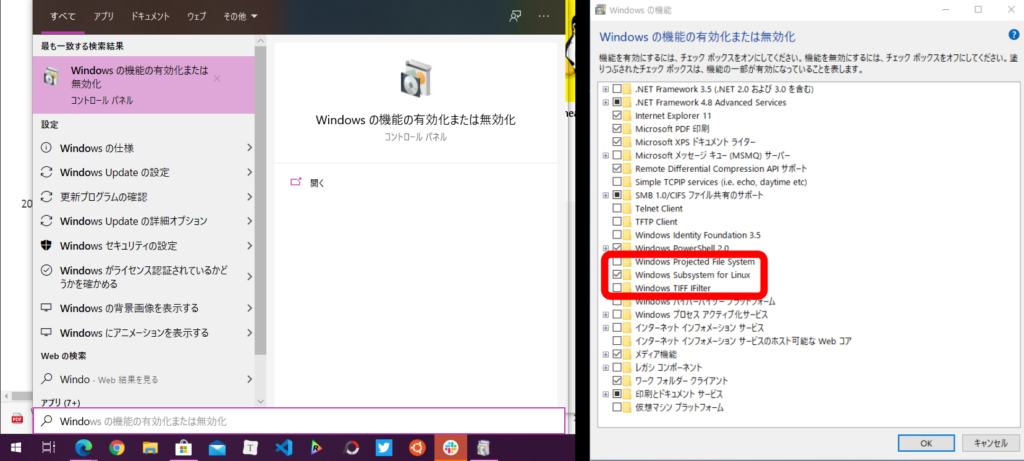
Windows 10 Homeの場合、WSLの機能がONになっていないので、「Windowsの機能の有効化と無効化」から「Windows Subsystem for Linux」にチェックマークを入れておきます。
Proの場合は検証していませんが、なしでもいけるのかもしれません。(のちにUbuntuが起動しない場合はこれを行ってください。)
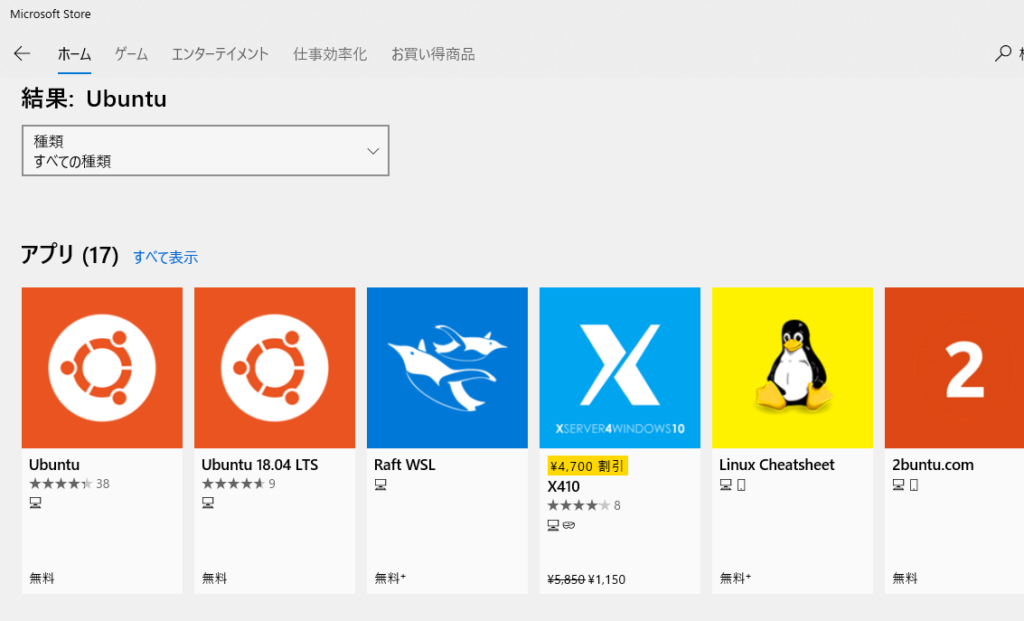
Ubuntu 18.04 LTSをインストールして起動。起動するとusernameとpasswordをインストールするように求められます。これは後で使うので覚えておいてください。
Ubuntu 18.04を起動すると、こうした感じのターミナルが表示されます。
最新のRとRStudio Serverのインストール
上記のサイトの手順ではRのバージョンが3.4.4になってしまうので、上の方法を踏まえつつ、以下の手順で最新版のRをインストールします。
echo -e "\n## For R package" | sudo tee -a /etc/apt/sources.list echo "deb https://cran.rstudio.com/bin/linux/ubuntu $(lsb_release -cs)-cran35/" | sudo tee -a /etc/apt/sources.list
まず、Ubuntu 18.04を起動して上記のコマンドでターミナルにsource.listに追加。
sudo apt-key adv --keyserver keyserver.ubuntu.com --recv-keys E298A3A825C0D65DFD57CBB651716619E084DAB9 #上記コマンドがプロキシやファイアウォールの設定などでうまく動かない場合は次の行を実行するらしいです gpg --keyserver hkp://keyserver.ubuntu.com:80 --recv-keys E298A3A825C0D65DFD57CBB651716619E084DAB9 gpg -a --export E298A3A825C0D65DFD57CBB651716619E084DAB9 | sudo apt-key add -
sudo apt update sudo apt install r-base
ここまでくるとRの3.6.3がインストールされているはず。
RStudio Serverのインストール
sudo apt-get install gdebi-core wget https://download2.rstudio.org/server/bionic/amd64/rstudio-server-1.2.1335-amd64.deb sudo gdebi rstudio-server-1.2.1335-amd64.deb
これでRStudio Serverのインストールは完了。
# 起動 sudo rstudio-server start # 終了 sudo rstudio-server stop
Ubuntuのターミナルで上記のコマンドをそれぞれ打つと起動と停止が行えます。ブラウザからlocalhost:8787にアクセスすると、RStudio Serverとご対面。ユーザー名とパスワードは、WSLでUbuntuを初めて立ち上げたときに決めたものになります。
終了する際は、データの保存をお忘れなく。
C:\Users\[ユーザー名]\AppData\Local\Packages\CanonicalGroupLimited.UbuntuonWindows_79rhkp1fndgsc\LocalState\rootfs\home
ちなみにRStudio Serverのディレクトリは、上記の場所らしい。
Stan・devtools等の準備
sudo apt-get install libcurl4-openssl-dev libssl-dev libxml2-dev build-essential g++ sudo apt update sudo apt upgrade sudo add-apt-repository ppa:marutter/c2d4u3.5 sudo apt install r-cran-tidyverse r-cran-devtools r-cran-rstan
CmdStanRを使うにあたりdevtoolsが必要なのですが、上記のコマンドをUbuntuのターミナルに打ち込みます。RStudioのパッケージ管理からdevtoolsをインストールしてもエラーが生じます。
CmdStanRの準備
devtools::install_github("stan-dev/cmdstanr")
library(cmdstanr)
install_cmdstan()
RStudioのコンソール上で、上記の3コードを実行。
CmdStanRを使う
CmdStanの準備
devtools::install_github("stan-dev/cmdstanr")
library(cmdstanr)
install_cmdstan()
cmdstanのインストールを上記のコマンドで。
set_cmdstan_path()
cmdstanのディレクトリをセット。
CmdStanでコンパイルと実行
file <- file.path("/home/(username)/(Folder_name)/hogehoge.stan")
mod <- cmdstan_model(file)
モデルのコンパイルに関しては上記の様に、ファイルパスを指定してから、cmdstan_modelでコンパイルします。
ファイルパスに関してですが、home/ユーザー名と順番に指定してあげるのが確実です。
#データはいつもどおりリスト形式 data_list <- list() # 実行はモデルに$とsample fit <- mod$sample( data = stan_data, seed = 1234, parallel_chains = 4, chains = 4, iter_warmup = 1000, iter_sampling = 1000 )
データはいつもどおりリスト形式。サンプリングに関しては上記のコマンドで実行する様になっています。RStanとは表記が違うところがあるのには注意!。
CmdStanでの結果
stanfit <- rstan::read_stan_csv(fit$output_files()) print(stanfit)
rstan::read_stan_csv関数を使って、CmdStanのオブジェクトを読み込むと、通常のRStan同様に結果が扱えるらしい。
参考にしたサイト



